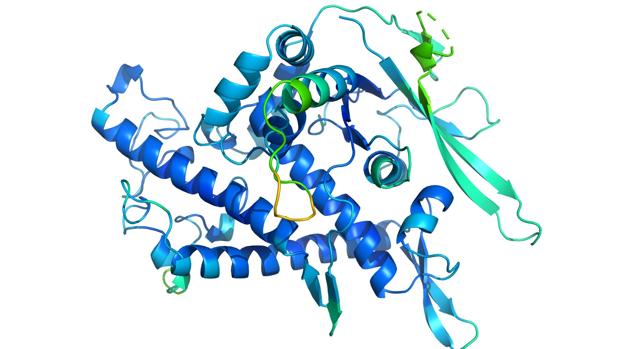
 Las proteínas no son Sólo nutrientes presentes en los alimentos. Consisten en largas y complejas moléculas, compuestas de aminoácidos, que llevan a cabo La mayor parte de las funciones en el interior de las células, De exactamente la misma forma que la replicación del material genético, la obtención de energía o la señalización de todas y cada una de las rutas indispensables para su funcionamiento. Una de las características más importantes de las proteínas es que su función depende de de qué manera se pliegan: sus propiedades físico-químicas las hacen adquirir una estructura tridimensional, sin la que no pueden funcionar. Por eso, Desde hace 50 años uno de los retos más importantes de la Biología ha sido precisamente averiguar de qué forma se pliegan las proteínas, lo que tiene infinitas aplicaciones en la investigación básica, en procesos industriales y en el sector del crecimiento de fármacos: por servirnos de un ejemplo, conocer cómo se pliega la proteína S del coronavirus es crucial para diseñar vacunas u otros medicamentos. Hoy se conocen alrededor de 180 millones de proteínas, Pero Solo se ha podido averiguar de qué forma se pliegan 170.000 de ellas. Esto se ha logrado A través de técnicas experimentales, Al igual que la cristalografía de rayos X, que las «congela» y cristaliza, para resolver su estructura con fotones, o bien la resonancia magnética. Pero, Al semejante que este trabajo es muy complicado y Algunas proteínas «se resisten», De la misma forma se trabaja en modelos y en aproximaciones de bioinformática para predecir el plegamiento de las proteínas Desde su secuencia de aminoácidos. Por desgracia, hay tantas posibilidades y dificultades en cada proteína que hasta en seguida no se ha logrado un gran desenvolvimiento en el campo. Todo esto parece haber cambiado. Esta semana, DeepMind, filial de Google que ha creado sistemas de Inteligencia Artificial (IA) capaces de aprender y vencer en el ajedrez, el go o bien en videojuegos de tiros, ha desarrollado otra IA, de nombre AlphaFold, capaz de predecir la estructura tridimensional de las proteínas. En concreto, ha logrado alcanzar una precisión del 92% en esta labor, en un encuentro bianual destinado a colocar a prueba a modelos bioinformáticos, y de nombre CASP, de «Critical Assessment of protein Structure Prediction». Sus resultados se han anunciado esta semana, No obstante todavía no se han publicado en una revista científica revisada por pares. Ajuste entre dos estructuras descubiertas experimentalmente (en verde) y predicha por AlphaFold (en azul) – DeepMind
Las proteínas no son Sólo nutrientes presentes en los alimentos. Consisten en largas y complejas moléculas, compuestas de aminoácidos, que llevan a cabo La mayor parte de las funciones en el interior de las células, De exactamente la misma forma que la replicación del material genético, la obtención de energía o la señalización de todas y cada una de las rutas indispensables para su funcionamiento. Una de las características más importantes de las proteínas es que su función depende de de qué manera se pliegan: sus propiedades físico-químicas las hacen adquirir una estructura tridimensional, sin la que no pueden funcionar. Por eso, Desde hace 50 años uno de los retos más importantes de la Biología ha sido precisamente averiguar de qué forma se pliegan las proteínas, lo que tiene infinitas aplicaciones en la investigación básica, en procesos industriales y en el sector del crecimiento de fármacos: por servirnos de un ejemplo, conocer cómo se pliega la proteína S del coronavirus es crucial para diseñar vacunas u otros medicamentos. Hoy se conocen alrededor de 180 millones de proteínas, Pero Solo se ha podido averiguar de qué forma se pliegan 170.000 de ellas. Esto se ha logrado A través de técnicas experimentales, Al igual que la cristalografía de rayos X, que las «congela» y cristaliza, para resolver su estructura con fotones, o bien la resonancia magnética. Pero, Al semejante que este trabajo es muy complicado y Algunas proteínas «se resisten», De la misma forma se trabaja en modelos y en aproximaciones de bioinformática para predecir el plegamiento de las proteínas Desde su secuencia de aminoácidos. Por desgracia, hay tantas posibilidades y dificultades en cada proteína que hasta en seguida no se ha logrado un gran desenvolvimiento en el campo. Todo esto parece haber cambiado. Esta semana, DeepMind, filial de Google que ha creado sistemas de Inteligencia Artificial (IA) capaces de aprender y vencer en el ajedrez, el go o bien en videojuegos de tiros, ha desarrollado otra IA, de nombre AlphaFold, capaz de predecir la estructura tridimensional de las proteínas. En concreto, ha logrado alcanzar una precisión del 92% en esta labor, en un encuentro bianual destinado a colocar a prueba a modelos bioinformáticos, y de nombre CASP, de «Critical Assessment of protein Structure Prediction». Sus resultados se han anunciado esta semana, No obstante todavía no se han publicado en una revista científica revisada por pares. Ajuste entre dos estructuras descubiertas experimentalmente (en verde) y predicha por AlphaFold (en azul) – DeepMind
«Hemos estado atascados con este problema —el de de qué manera se pliegan las proteínas— En medio casi 50 años», ha explicado en un parte John Moult, presidente y cofundador de CASP, e investigador en la Universidad de Maryland (EE.UU.). «Ver de qué manera DeepMind ha creado una solución para esto (…) es un instante muy especial». No Se trata de un avance menor. Conforme ha informado «Nature.com», poder predecir la estructura de una proteína, A partir de su secuencia de aminoácidos sería un grandísimo salto para las ciencias de la vida y la Medicina. Aceleraría enormemente los esfuerzos para comprender los ladrillos básicos de la vida y haría que la investigación de nuevos medicamentos fuera más rápida y avanzada. Según ha expresado Demis Hassabis, director general de DeepMind: «Creo que ésta es la cosa más significativa que hemos logrado, en términos del impacto que tendrá en el mundo real». «Creo que ésta es la cosa más significativa que hemos logrado, en términos del impacto que tendrá en el mundo real» «Es un avance de primer mandato, Sin duda uno de los resultados científicos más importantes que he presenciado en mi vida», ha comentado para «Nature» Mohammed AlQuraishi, biólogo computacional de la Universidad de Columbia, y participante en el CASP. Tanto que, al haberse resuelto el problema fundamental, ha expresado que muchos Grupos se dedicarán a otra cosa. Una antigua búsqueda En 1972, el Nobel en Química Christian Anfinsen postuló que la estructura de una proteína está completamente determinada por su secuencia de aminoácidos. Sin embargo en 1969, Cyrus Levinthal predijo que haría falta más tiempo que la edad del universo para enumerar todas y cada una de las posibles configuraciones de una proteína típica, por medio de cálculos —predijo que una proteína típica tiene 10^300 conformaciones—. Curiosamente, A pesar de ese número de configuraciones, las proteínas se pliegan a medida que se van generando, en los ribosomas, en cuestión de unos pocos milisegundos. En 1994 se fundó el CASP para acelerar la investigación y colocar en común los progresos. Desde entonces, cada un par de años, en este partido se han seleccionado proteínas recientemente determinadas por métodos experimentales, para poner a prueba modelos bioinformáticos predictivos, sin que los desarrolladores pudieran saber cuál era la estructura de la proteína analizada. Para medir el logro de sus esfuerzos, se desarrolló una medida, famosa De este modo como GDT (de Test de Distancia Global, en inglés), cuya puntuación va de cero a 100: aquellas puntuaciones cercanas a 90 suelen ser consideradas Así tal y como una estimación competente. En el undécimo Cuarto partido de CASP la inteligencia artificial AlphaFold ha logrado una puntuación GDT media de 92,4, Tras trabajar con Múltiples proteínas. Esta precisión se traduce en que la IA ha cometido un fallo comparable al ancho de un átomo o 0,1 nanómetros, en el horario en que un nanómetro es la millonésima comunicado de un milímetro. Una auténtica revolución
«Estos resultados abren la puerta a que los biólogos usen la predicción computacional de la estructura Del mismo modo que herramienta nuclear en la investigación científica», han explicado Desde DeepMind. «Nuestros métodos pueden ser especialmente útiles para importantes tipos de proteínas, De la misma forma que las de membrana —son las que se sitúan en la bicapa lipídica que separa el interior de las células del medio exterior— que son especialmente difíciles de cristalizar y, en consecuencia, de determinar experimentalmente». «Este trabajo computacional es un avance maravilloso en el problema del plegamiento de las proteínas, un enorme reto para la Biología A partir de hace 50 años» «Este trabajo computacional es un avance maravilloso en el problema del plegamiento de las proteínas, un enorme reto para la Biología A partir de hace 50 años», ha explicado Venki Ramakrishnan, presidenta de la Royal Society. «Y ha ocurrido décadas Antes de lo cual muchos habían predicho. Va a ser muy emocionante ver las muchas formas De exactamente la misma forma que esto varía esencialmente la investigación biológica». «Lo que el elenco de DeepMind ha logrado hacer es fabuloso y cambiará el futuro de la biología estructural y la investigación de proteínas», ha explicado en «Sciencemag.org» Janet Thornton, directora emérita del Instituto Europeo de Bioinformática. Para conseguir estos resultados, el club de DeepMind ha estado cuatro años trabajando en crear y entrenar a una red neural capaz de procesar «gráficas espaciales», que representan el plegamiento de las proteínas y las relaciones de sus residuos de aminoácidos. Dicho sistema de IA aprende a refinar estas gráficas empleando secuencias relacionadas, alineamientos de Varios secuencias y otras representaciones. Proceso de aprendizaje para procesar gráficas espaciales y predecir la estructura 3D de proteínas – DeepMind
Después de varias iteraciones, el sistema aprendió a hacer predicciones acerca de la estructura física con enorme precisión. Para ello, se dirigió preciso entrenarlo con la secuencia de las 170.000 proteínas conocidas y aprovechar También grandes bases de data de proteínas desconocidas, usando semanas de computación en un número de procesadores comparable a 100 o 200 GPUs o unidades de preocesamiento gráfico. Además fue preciso crear «algoritmos de tensión», que permitieron conectar pequeños Grupos de aminoácidos, Ya antes de formar un grupo, De exactamente la misma forma que si es que se tratara de un puzzle resuelto Desde pequeños Grupos. Después, los estudiosos de DeepMind están trabajando para publicar sus avances en una gaceta científica y en localizar nuevas formas de facilitar el acceso a esta herramienta gran escala. Asimismo, pretenden averiguar cómo estas predicciones pueden participar a estudiar Ciertas enfermedades, a facilitar el desarrollo de medicamentos y complementar métodos experimentales ya existentes. No obstante, AlphaFold todavía no es perfecto. Por servirnos de un ejemplo, tiene problemas con estructuras formadas por la repetición de pequeños segmentos, y todavía no deja estudiar complejos de Varios proteínas que desempeñan juntas su función en las células. Un futuro prometedor
Esta IA no es Sólo un potente «telescopio» con el que adentrarse en el desconocido universo de millones de proteínas cuya estructura no se ha revelado. Con la vista puesta en el futuro, los investigadores de DeepMind han comentado que esta herramienta puede ser útil para responder a próximas pandemias, Ya que AlphaFold pudo predecir con triunfo las estructuras de ORF8 y ORF3a, dos proteínas del SARS-CoV-2. Todavía más allá, han sugerido que la IA podrá ser de ayuda para estudiar de qué manera las proteínas interaccionan con el ADN, el ARN u otras moléculas. «Los sistemas De exactamente la misma manera que AlphaFold demuestran el espectacular potencial de las IAs Del mismo modo que herramienta para avalar descubrimientos fundamentales», han concluido. «(…) Hay muchos aspectos de nuestro universo que son desconocidos. El avance anunciado después nos da más seguridad de que la IA se convertirá en una de las herramientas más útiles para expandir las fronteras del conocimiento científico».
